Kit para la determinación del número de repeticiones del triplete GAA del gen FXN mediante el análisis de fragmentos fluorescentes.
Información del producto
La Ataxia de Friedreich (FRDA) es la Ataxia hereditaria más común, tratándose de una enfermedad autosómica recesiva degenerativa.
La anomalía genética asociada con la FRDA es la expansión del triplete de repetición GAA localizada en el primer intrón del gen que codifica la frataxina (FXN). Los rangos patológicos de los alelos con expansión del triplete GAA están en un rango de repeticiones entre 50 y 1300, de manera que existen 3 intervalos diferentes:
- Individuos sanos (5-30 repeticiones)
- Pacientes con síntomas leves (30-49 repeticiones)
- Pacientes con síntomas severos (50-1300 repeticiones).
Uso previsto
Adellgene® Friedreich’s Ataxia es un kit de diagnóstico in vitro semiautomatizado de uso en laboratorios clínicos diseñado para la determinación cuantitativa del número de repeticiones del triplete GAA (guanina-adenina-adenina) presentes en el primer intron del gen que codifica la frataxina (FXN) localizado en el cromosoma 9, que puede desembocar en la aparición de ataxia de Friedreich. Esta determinación pretende ayudar en el diagnóstico asociado con la enfermedad, cuyos hallazgos clínicos abarcan desde síntomas leves hasta graves.
Este kit permite la identificación tanto de alelos sanos que tienen entre 5 y 30 repeticiones GAA, pacientes con fenotipo leve (30-49 repeticiones), y severos (50-1300).
La determinación se basa en la reacción en cadena de la polimerasa (PCR) del ADN genómico extraído a partir de sangre periférica seguido de un análisis del tamaño de los fragmentos fluorescentes amplificados en un analizador genético y conversión de dicho tamaño en su respectivo número de repeticiones GAA.
Los pacientes que pueden beneficiarse de esta determinación son los remitidos por un especialista. El usuario previsto de este kit es personal técnico entrenado y cualificado para realizar el protocolo descrito en las instrucciones de uso y la interpretación de sus resultados.
Flujo de trabajo

Resultados
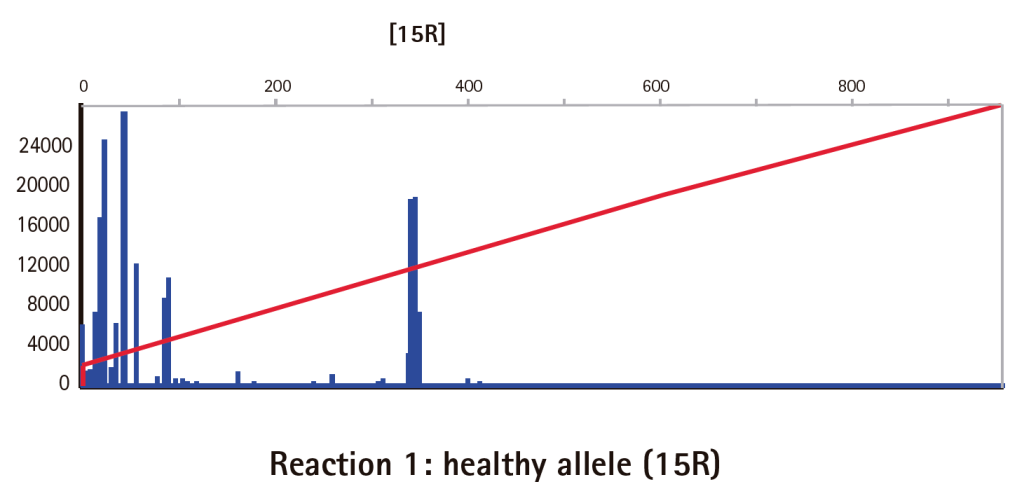
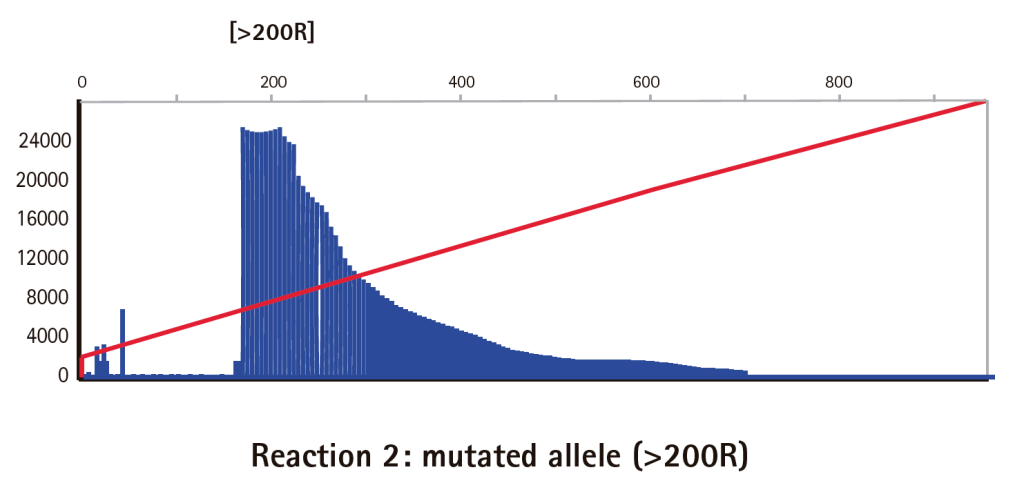

Limitaciones
- Las mutaciones (mutaciones puntuales, inserciones, deleciones) en los sitios de hibridación de los primers pueden dar lugar a la falta de definición de alelos. Otras tecnologías podrían ser necesarias para resolver el genotipado.
- Los datos y la interpretación de los resultados deben ser revisados por personal cualificado.







